温馨提示
邮箱 galaxy@ribobio.com 专门用于接收本平台所有用户在使用及注册过程中遇到的相关问题,我们承诺不会通过此邮箱向任何用户主动发送任何信息。
1. 简介¶
Galaxy 是一个提供执行交互式分析的分析环境,同时确保结果分析具有透明和可重复性的在线生物信息分析平台,其在后台封装了很多高端的生物信息分析计算工具,并且通过网页以直观的使用界面方式把这些工具友好地可视化展示给用户,同时把计算和存储管理的细节隐藏起来,因此它消除了在执行许多常见类型的大规模分析过程中对专门的信息学专业知识的需求,使得 Galaxy 生物信息分析平台成为了目前生物医学研究领域最受欢迎的在线生物信息分析工具。
使用提醒
为提供更好的使用体验,推荐在 Chrome/Edge/Firefox 等主流浏览器下使用本平台,建议使用 Chrome。
本文档为定制化的 RiboBio Galaxy 平台(http://galaxy.ribobio.com:8088/)使用说明文档,旨在帮助更多的用户熟悉 RiboBio Galaxy,利用 RiboBio Galaxy 平台实现生物信息常见的一些分析,为售后提供支持与帮助。
RiboBio Galaxy 平台主要分为三大块内容:工具栏、参数设置栏、历史栏。

- 工具栏:位于界面左侧,主要包括本平台所有可用的分析工具集。
- 参数设置栏:位于界面中间,是对选定的某一个特具体工具进行参数设置,提交分析任务操作的详情页。
- 历史栏:位于界面右侧,是对当前用户执行分析的历史记录,用户可以通过该历史记录查看分析的具体信息、查看分析结果,以及对分析结果进行可视化处理等操作。
2. 注册登录¶
RiboBio Galaxy 默认是需要每个用户注册后才能登录使用。
2.1 注册¶
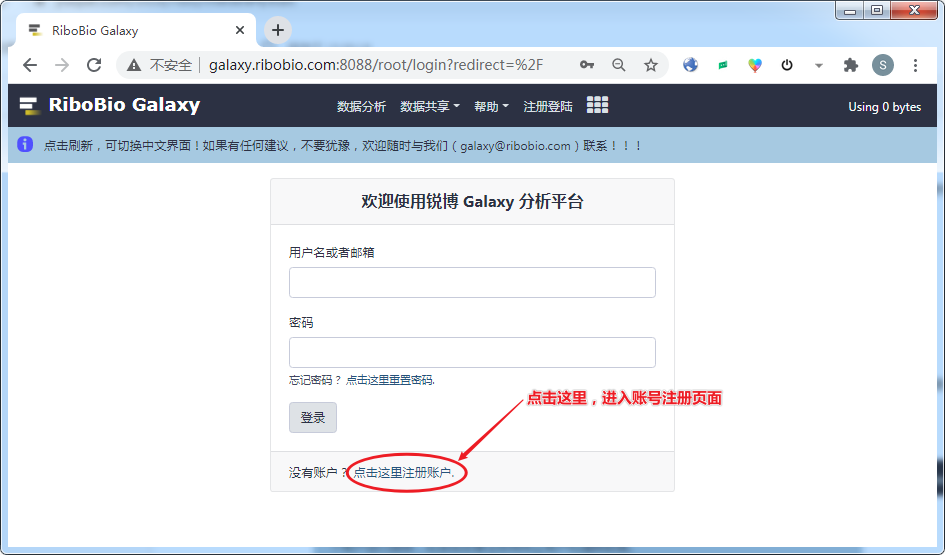
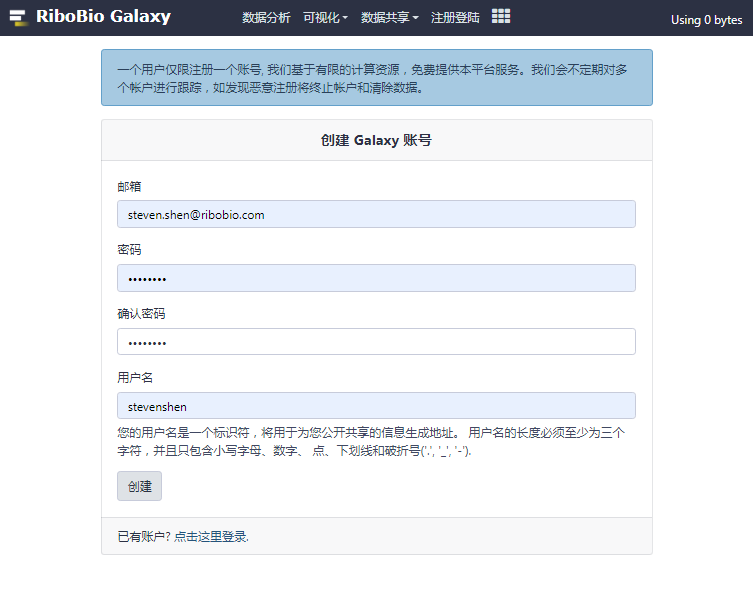
没有注册的用户,可以直接访问 RiboBio Galaxy 的链接,在注册页面中输入邮箱、密码、用户名进行注册。
2.2 激活¶
从 2024-10-25 起,RiboBio Galaxy 平台的所有注册用户统一调整为由管理员进行激活。用户账号注册完成后,需要:
-
通过注册邮箱向 galaxy@ribobio.com 发送一封账号激活邮件,邮件标题:《RiboBio Galaxy 账号激活》。
-
管理员收到激活邮件后,对应的用户账号 默认在 1 到 3 个工作日内完成激活。
特别提醒
对于账号没有进行激活的注册用户,您可以查看 RiboBio Galaxy 所有可用的工具和内容,但是无法上传数据和运行分析任务,您只有在验证电子邮件地址后才能上传数据或提交分析任务!
2.3 密码重置¶
用户如果忘记了密码,可以通过注册邮箱向 galaxy@ribobio.com 发送一封账号重置邮件,邮件标题:《RiboBio Galaxy 账号密码重置》。
管理员收到邮件后,将在 1 到 3 个工作日内完成对应账号密码重置,默认重置密码:galaxy@2024 。
特别提醒
密码重置后,用户及时应该登录平台通过 "账号管理" -> "偏好" -> "更改密码" 进行密码更改,以确保账号安全性!
3. 分析工具¶
RiboBio Galaxy 分析工具目前主要包括 获取数据、文本处理、(文本)过滤与排序、(生物信息数据)格式转换、绘图与数据可视化、RNA-seq 差异表达分析 等,为生信项目分析中最常见的售后相关问题提供了解决方案。
3.1 共享与示例数据¶
RiboBio Galaxy 在导航栏的 "数据共享" → "数据库" 内,为各个在线的工具提供了示例数据。
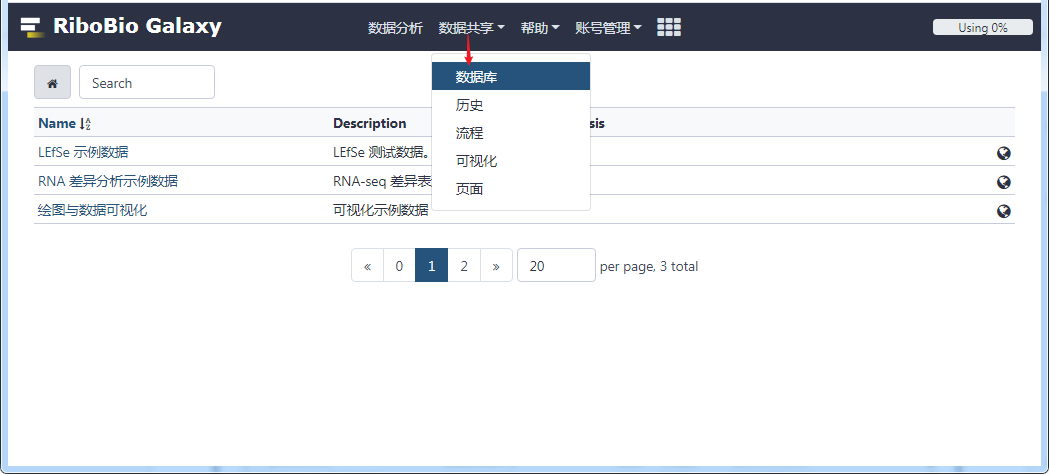
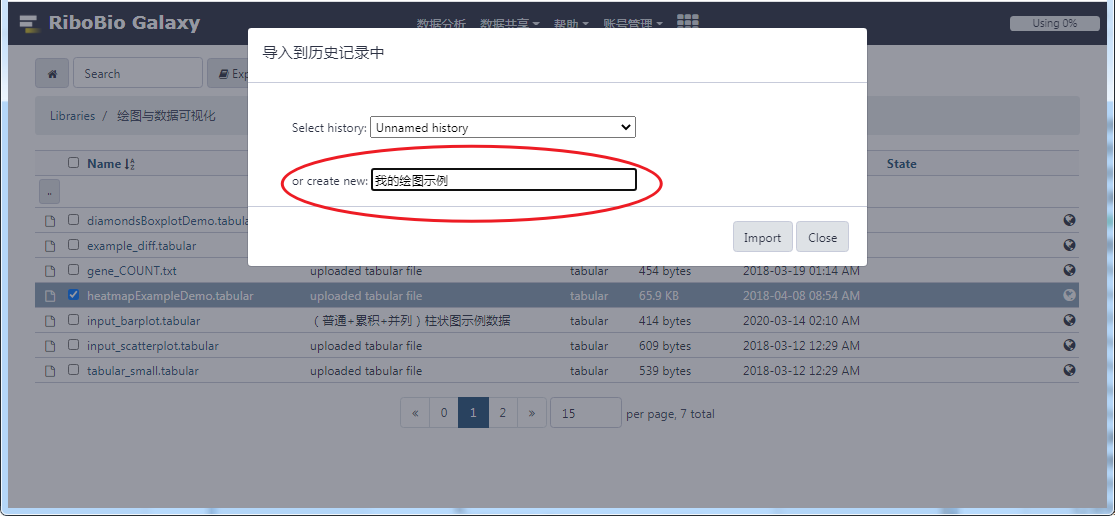
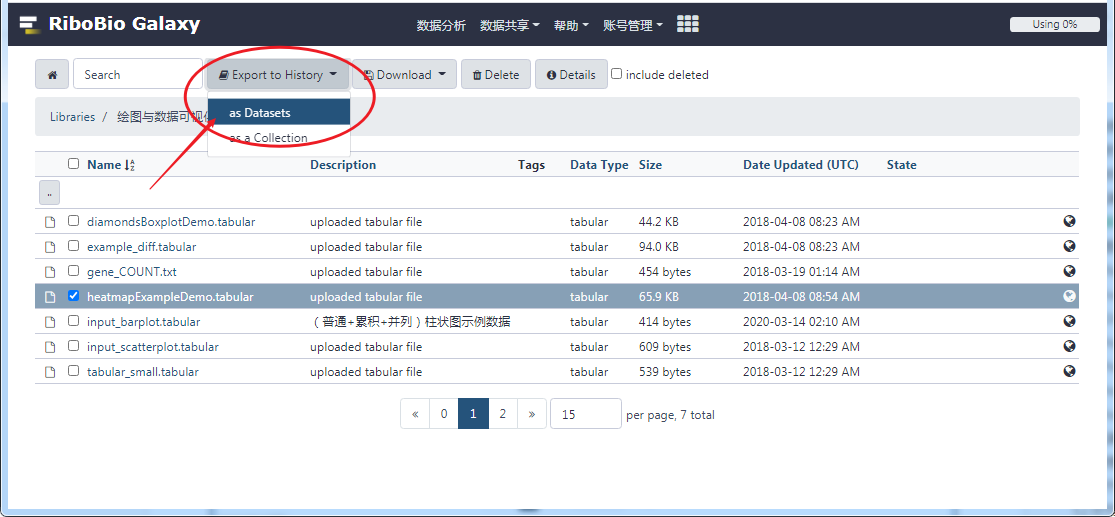
我们进入其中一个数据库(如,绘图与数据可视化)中,选中特定的数据后,"Export to History" → "as Datasets" 即可把数据库中共享的数据导入到历史记录中进行分析。
3.2 绘图与可视化¶
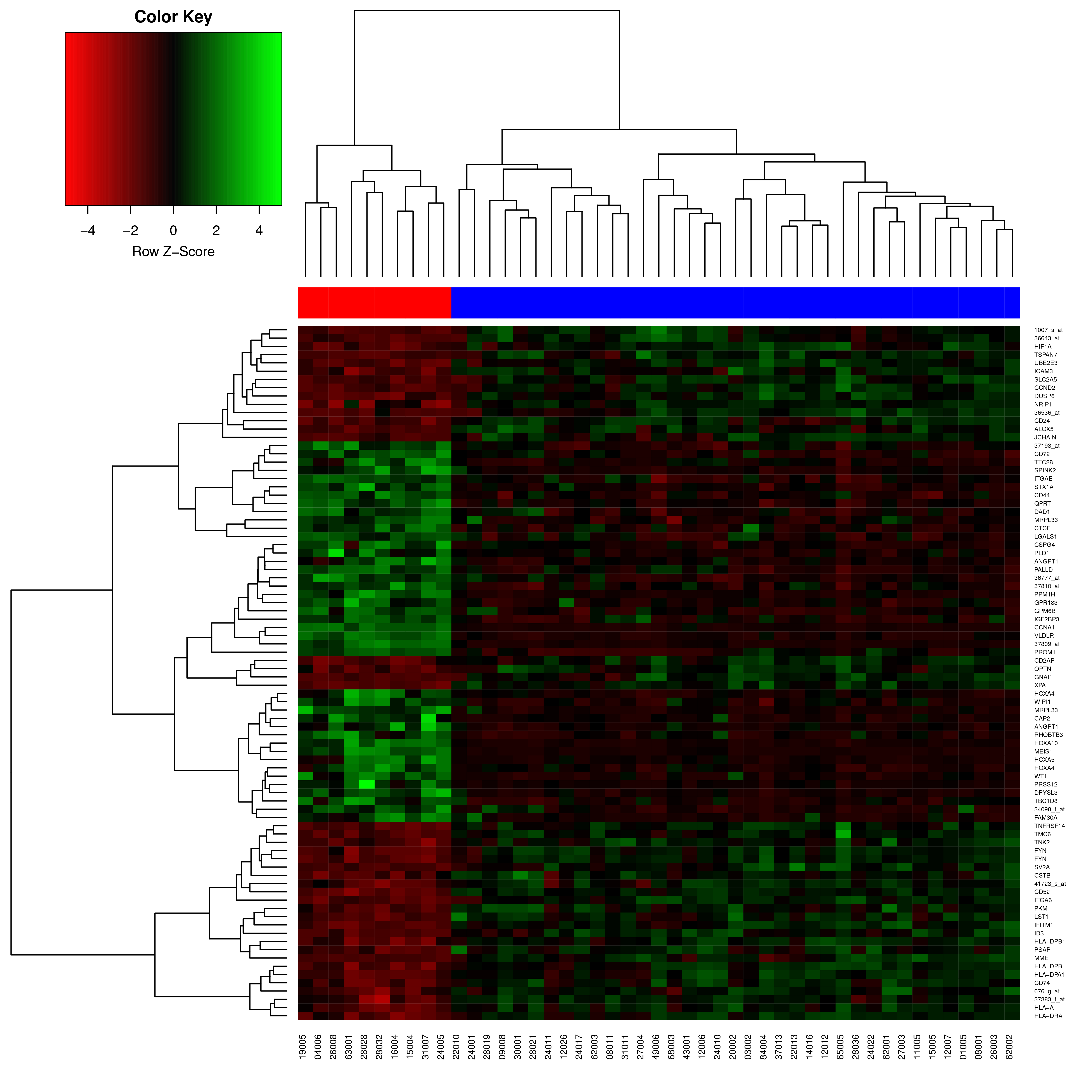
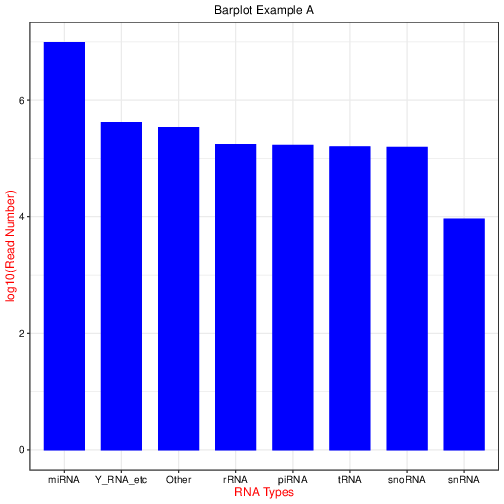
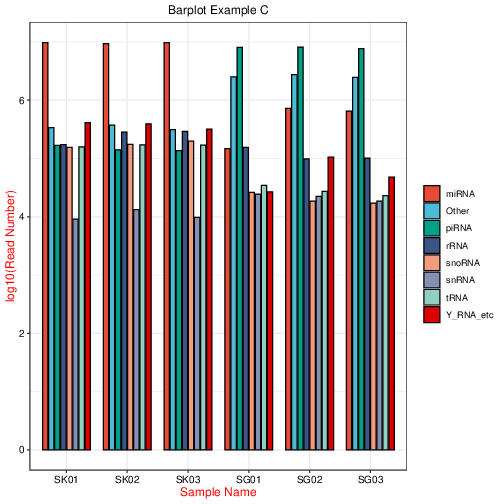
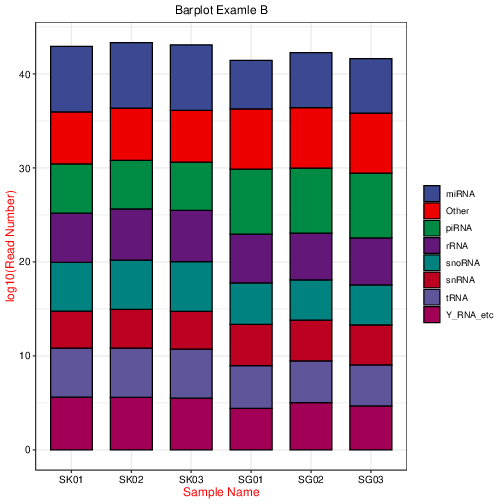
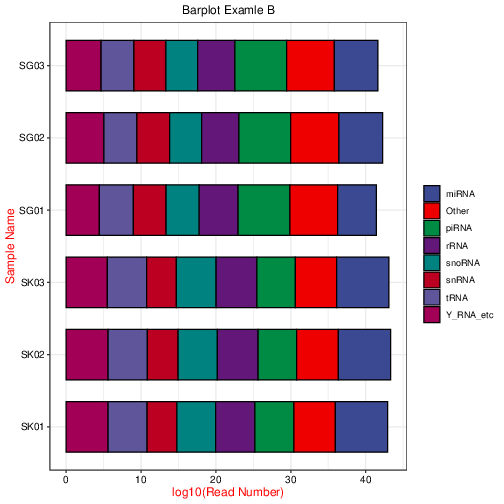
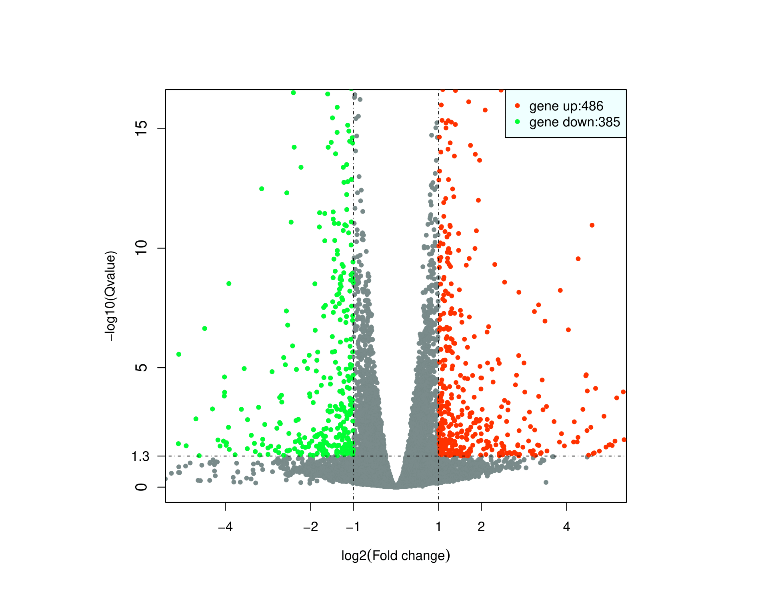
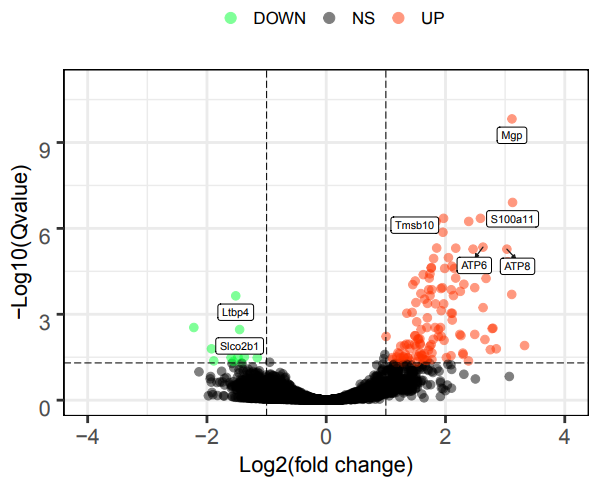
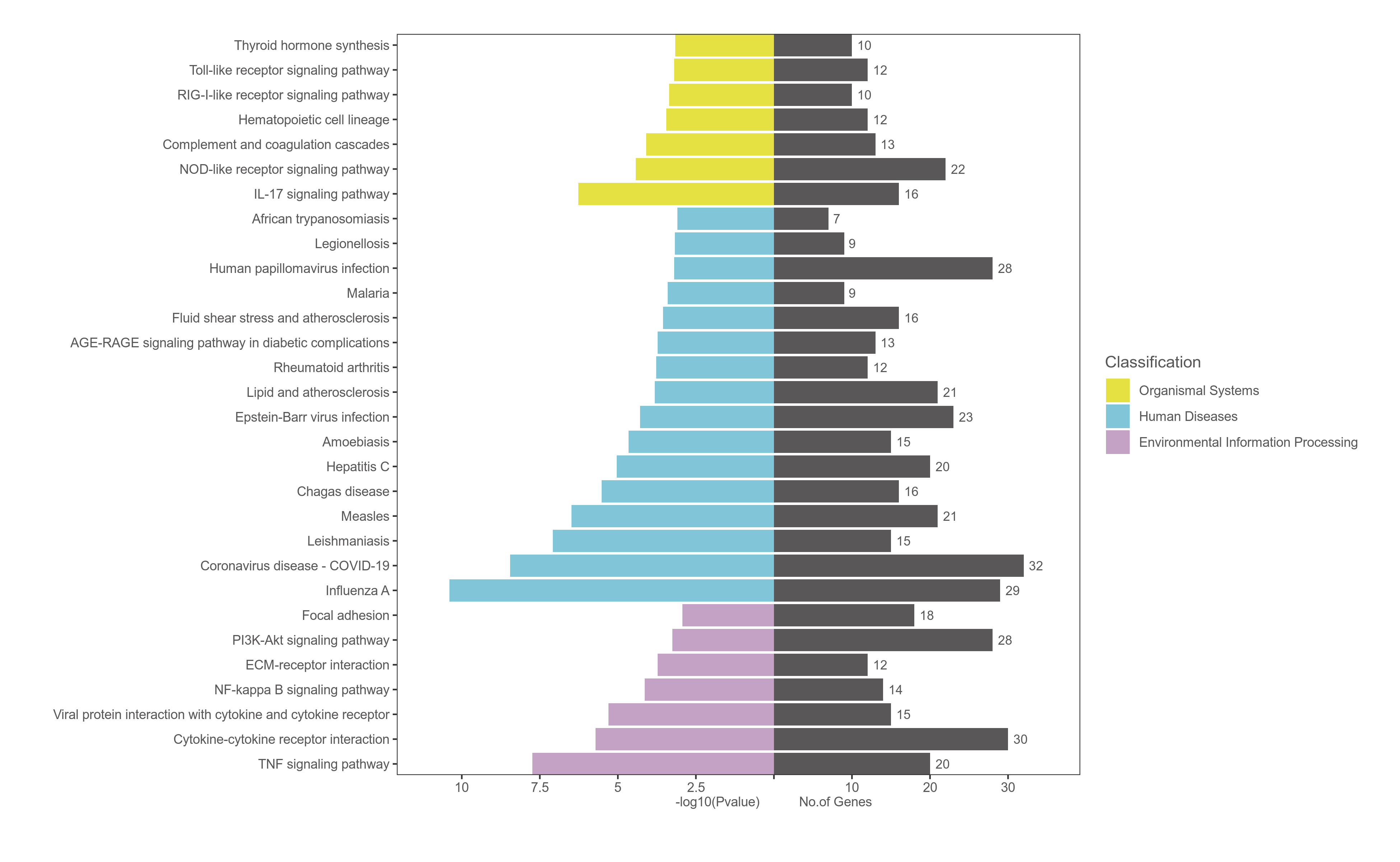
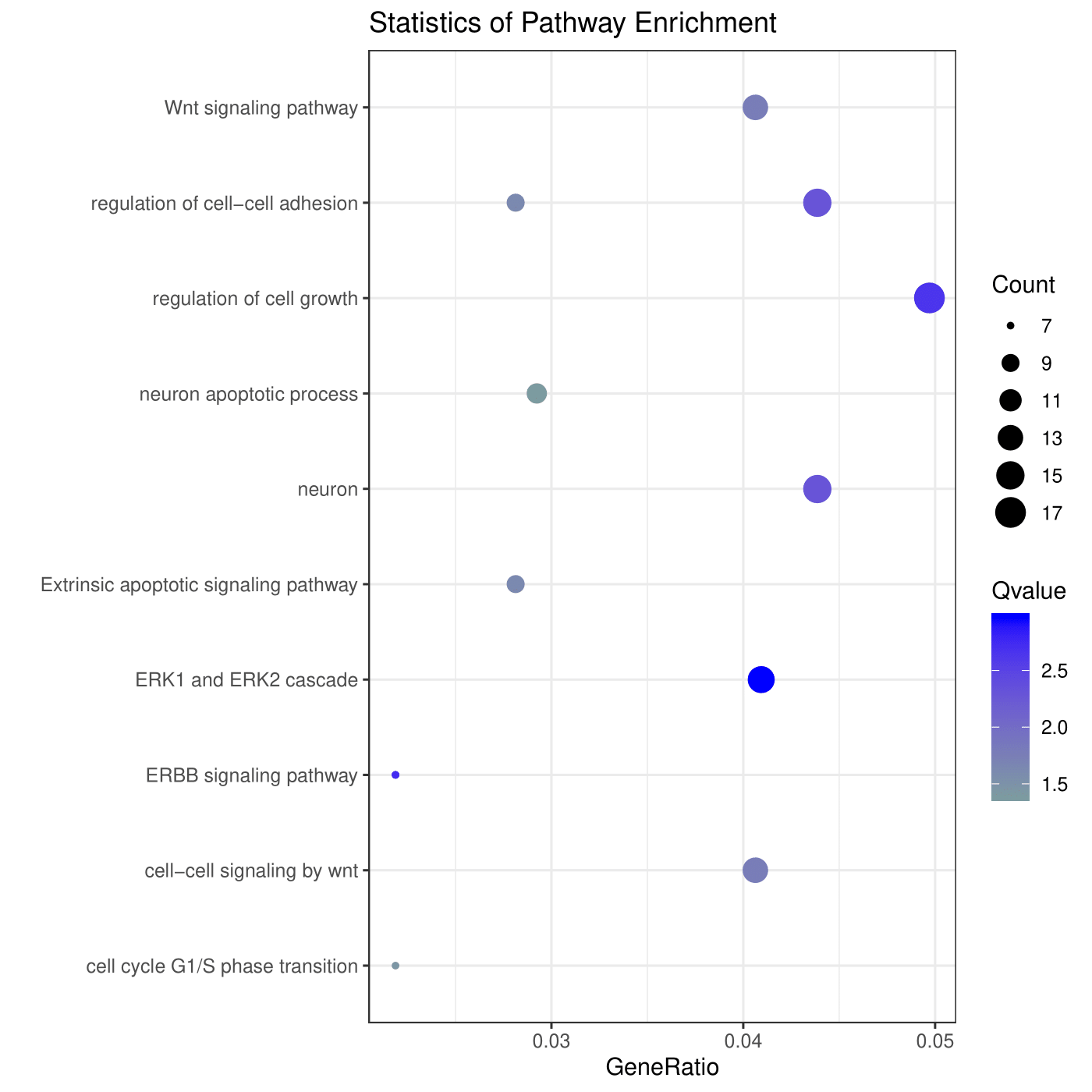
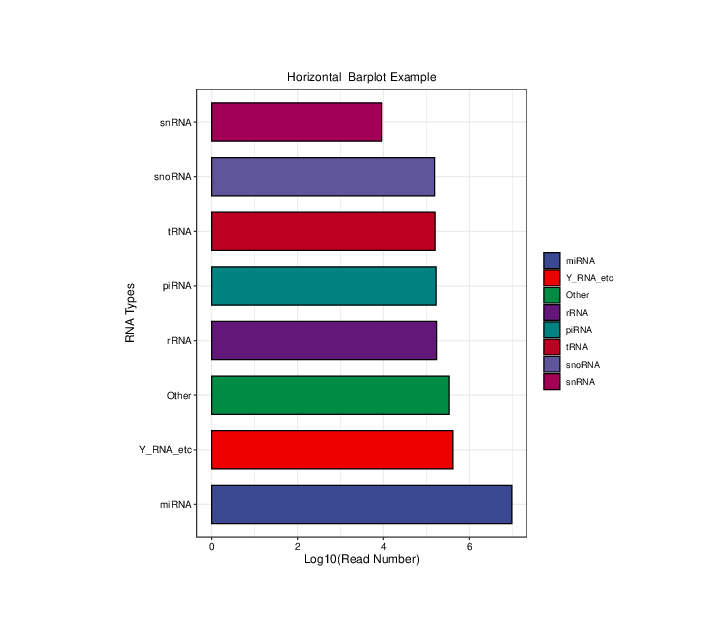
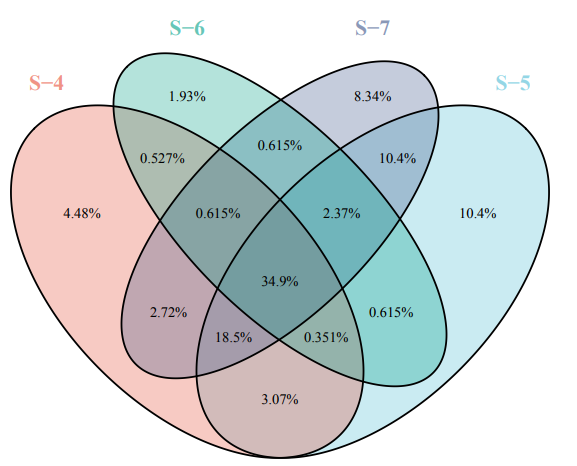
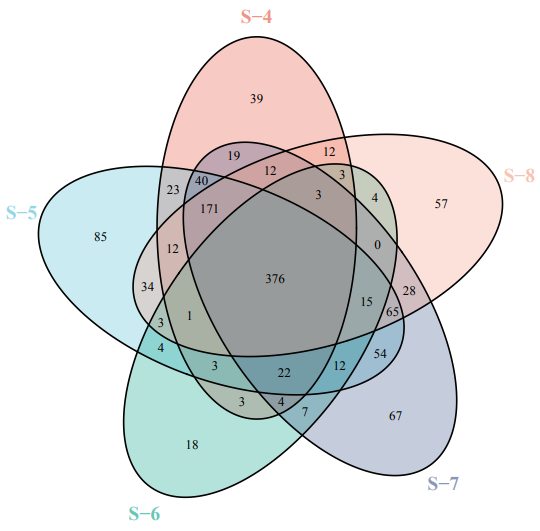
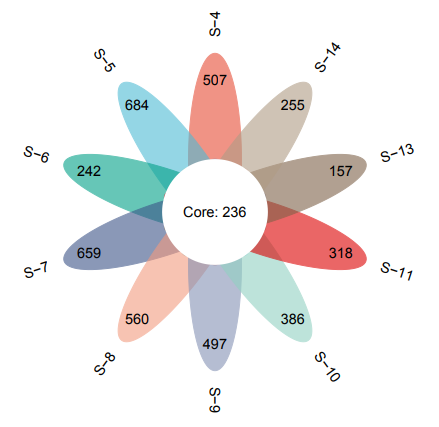
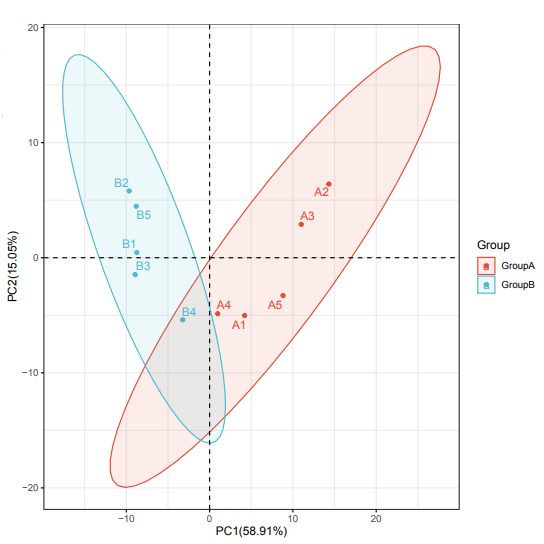
RiboBio Galaxy 中的绘图与数据可视化整合了目前常见热图(普通热图、环化热图)、柱状图(常规柱状图、累堆叠柱状图、并列柱状图)、散点图、火山图、韦恩图等生物信息分析中最长见的绘图类型。
RiboBio Galaxy 在每个工具的参数设置栏下面,都增加了对当前工具的详细说明和使用示例,在这个帮助文档里我们提供了当前工具的测试数据、参数设置示例,以及绘图结果展示,每个用户在使用该工具前,请先仔细阅读这一部分的内容,再按照指定的要求去上传和分析数据。
3.2.1 绘图总览¶
3.2.2 使用步骤¶
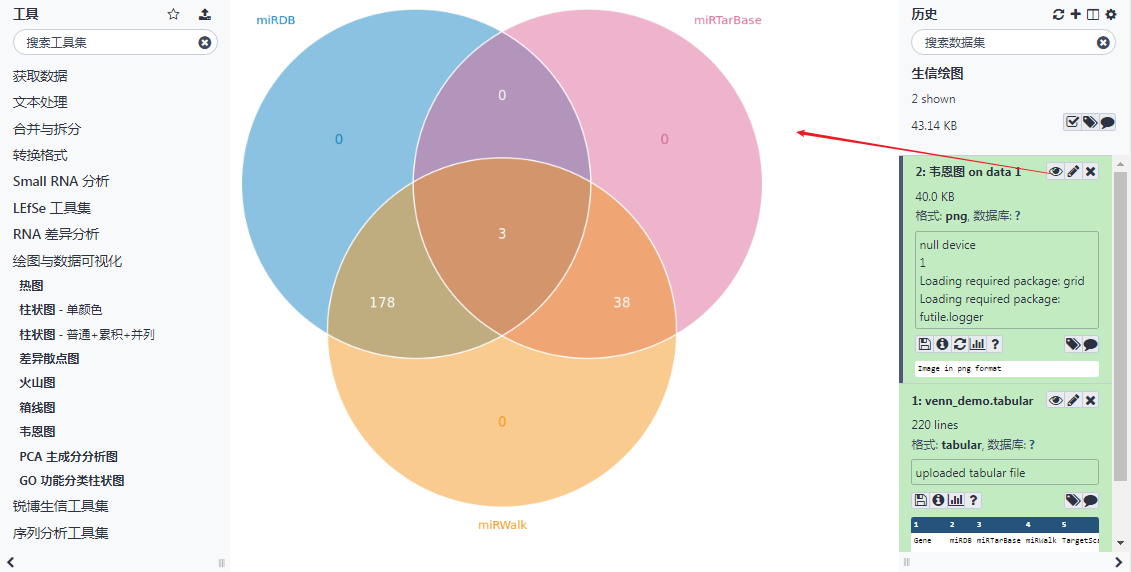
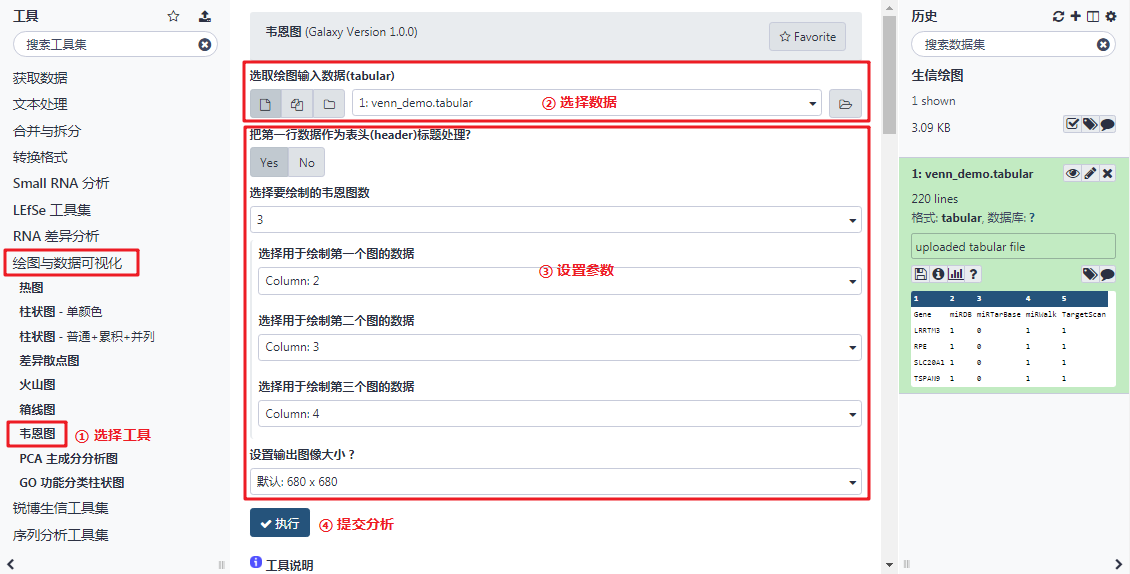
以韦恩图为例。RiboBio Galaxy 的韦恩图可以实现二元、三元和四元韦恩图的绘制,每一种韦恩图的绘制方法可以参考该工具参数页底部的说明文档。
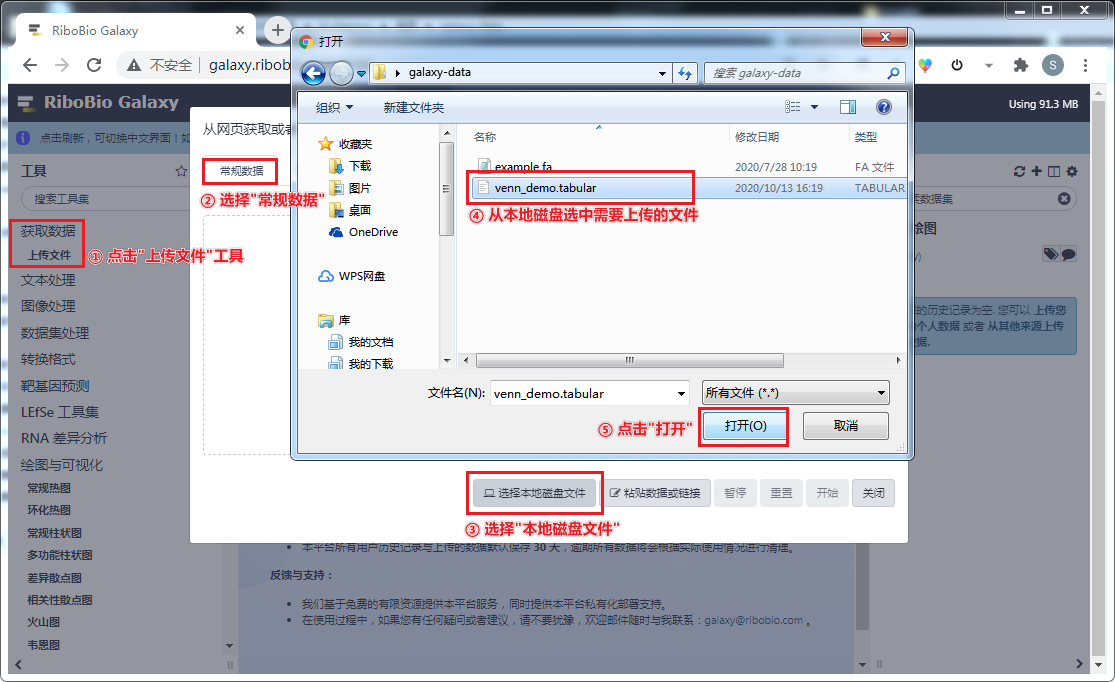
1. 获取数据
以 RiboBio Galaxy "绘图与数据可视化" 工具集下 "韦恩图" 提供的示例数据为例:http://galaxy.ribobio.com:8088/static/test-data/venn_demo.tabular。
点击 Galaxy 左边工具栏 "获取数据" → "上传文件" → "选择本地磁盘文件" → 选择你要上传的文件 → 设置 Type: tabular → 点击 "开始",完成最后的上传。
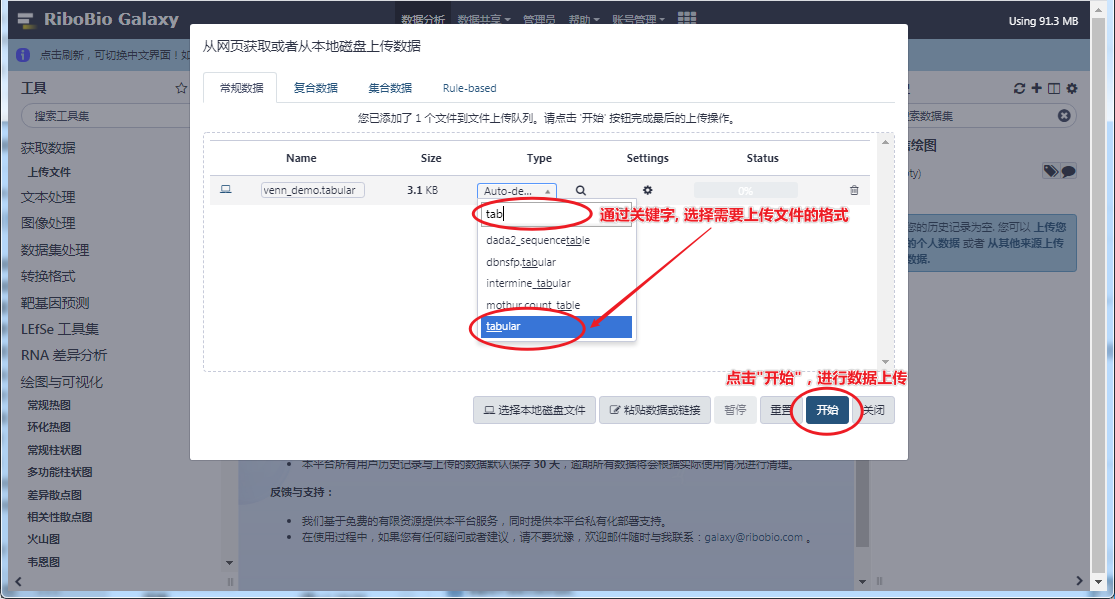
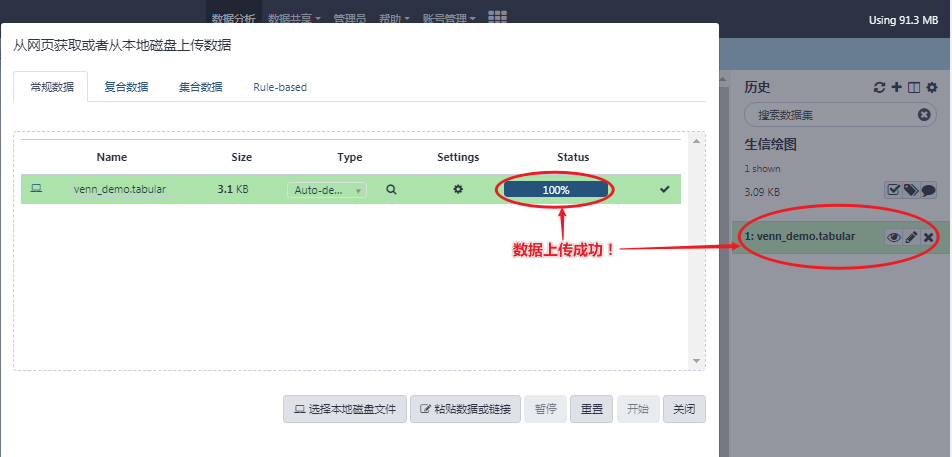
Tabular 数据格式
RiboBio Galaxy 中 绘图与数据可视化 的大部分工具都是基于 tabular 格式进行处理的,这其实就是一个 tab 制表符分隔的 txt 文件。
文件上传后,可以在右侧的历史栏中通过点击眼镜图片查看详细的数据内容。
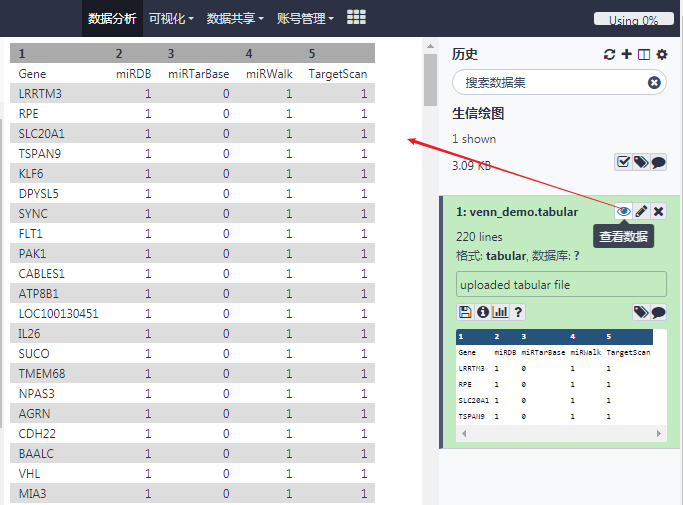
2. 选择工具,设置参数
点击左侧工具栏下 "绘图与数据可视化" → 选择"韦恩图",在中间栏出现的参数设置页选择需要执行韦恩图处理的数据,并设置参数,提交任务执行。
3. 查看结果
3.3 RNA 差异分析¶
RNA-seq 差异表达分析利用了 R 的 DESeq、DESeq2、edgeR、DEGseq 四个包,对 RNA-seq ( Gene、Transcript、mRNA、ncRNA ) 表达数据进行差异分析。
1. 输入数据¶
RNA-seq 差异表达分析示例数据,参考 http://galaxy.ribobio.com:8088/static/test-data/rna_seq_expr.tabular,该数据要求使用 TAB 制表符分隔,行为基因,列为样品分组,值为各个基因在不同分组中的表达值。
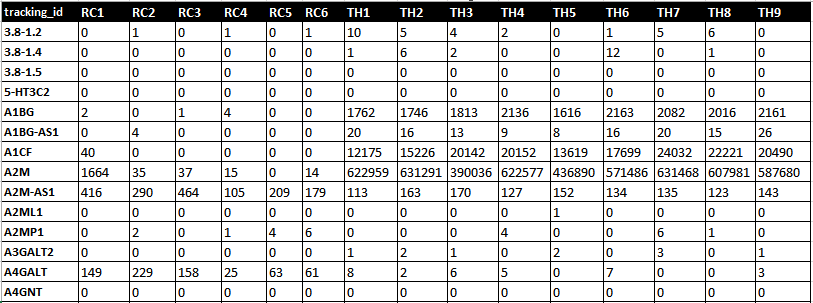
2. 参数设置¶
-
分组信息:
两两分组之间使用双下划线( __ )分隔,每个分组必须遵循以下要求:
格式:组名1:列名1,列名2,列名3,...__组名2:列名1,列名2,列名3,...
示例:Control1:TH1,TH2,TH3--Test1:TH4,TH5,TH6__Control1:TH1,TH2,TH3--Test2:TH7,TH8,TH9 -
差异表达计算方法:
请参考工具的页面说明。 -
log2( Fold_change ) 筛选值:
Fold_change 是样本质检表达量的差异倍数,这里取 log2(即以 2 为底 Fold_change 的对数)以缩小差异特别大和差异比较小的数值之间的差距。 -
Qvalue:
Q值,统计差异的显著性。 -
数据类型:
输入数据的数据类型( gene/transcript/mRNA/ncRNA )。
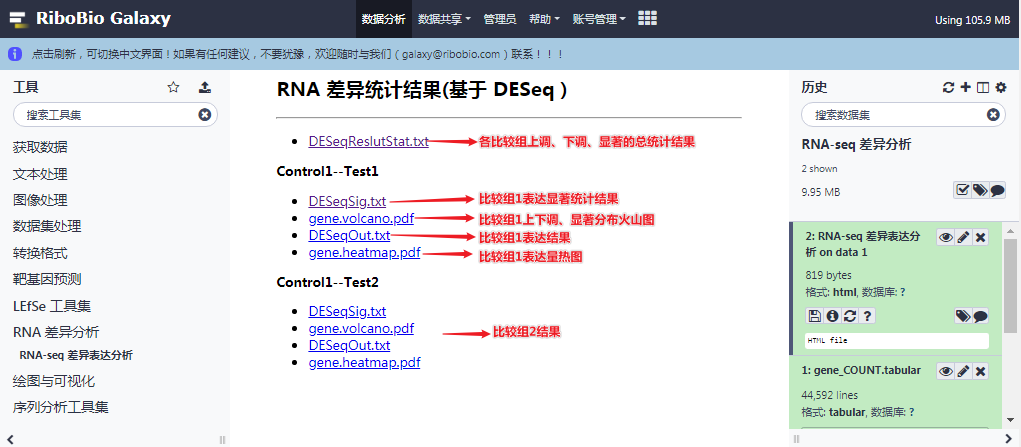
3. 结果说明¶
RNA-seq 差异表达分析 工具工具运行结束后,将得到一个 HTML 格式的结果列表。
3.4 文本处理¶
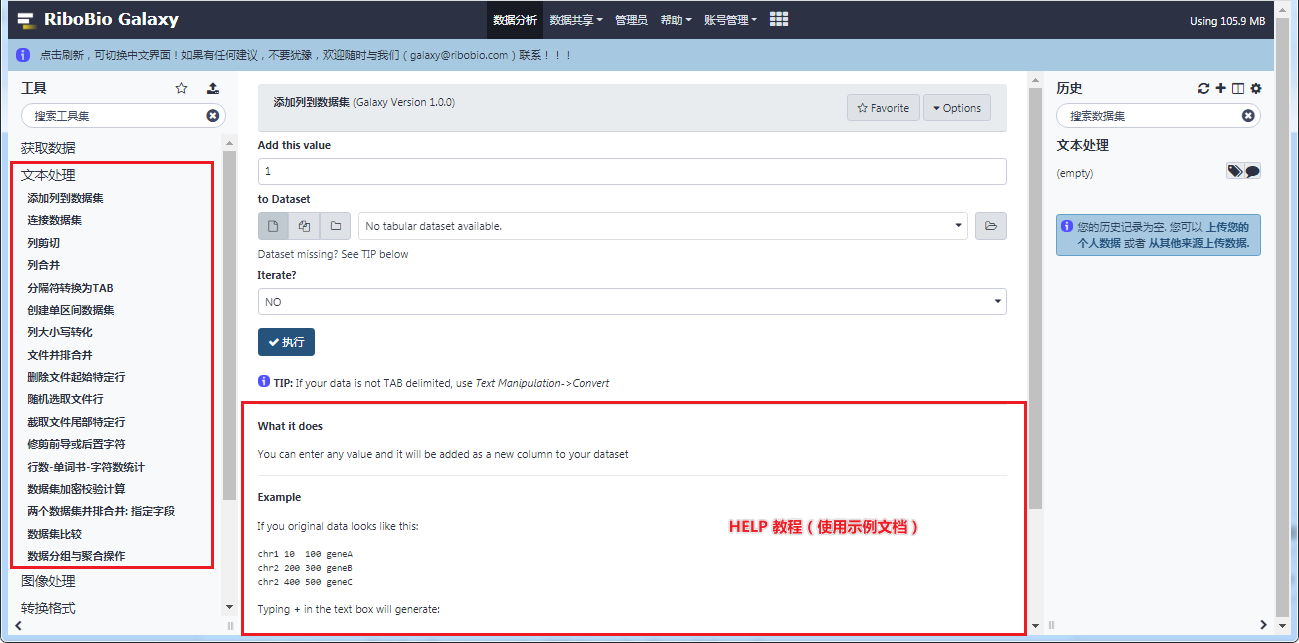
RiboBio Galaxy 把一些常用的文本数据处理,例如行列筛选、大小写转化、数据集比较,等等,进行了整合,并且 在每个文本处理的工具页面提供了详细的使用示例,以供大家使用参考。
3.5 LEfSe 分析¶
温馨提示
从 2024-12-01 起,本平台不再提供 LEfSe 分析相关服务,如有使用需要可以参考 Huttenhower Lab Galaxy Server 2.0 平台。
LEfSe (Linear discriminant analysis Effect Size) 是一种用于发现和解释高维度数据生物标识(基因、通路和分类单元等)的分析工具,可以进行两个或多个分组的比较,它强调统计意义和生物相关性,能够在组与组之间寻找具有统计学差异的生物标识(Biomarker)。
RiboBio Galaxy 已经集成了 LEfSe 的分析,下面简单介绍一下这个工具的使用。

下面我们来介绍一下如何在 RiboBio Galaxy 使用 LEfSe 进行分析。本使用示例章节仅对 RiboBio Galaxy 上的 LEfSe 的操作使用进行说明,不涉及具体选项的生物学解释,如要了解详细意义请参考相关页面说明。
本章节所使用示例数据:hmp_aerobiosis_small.tabular
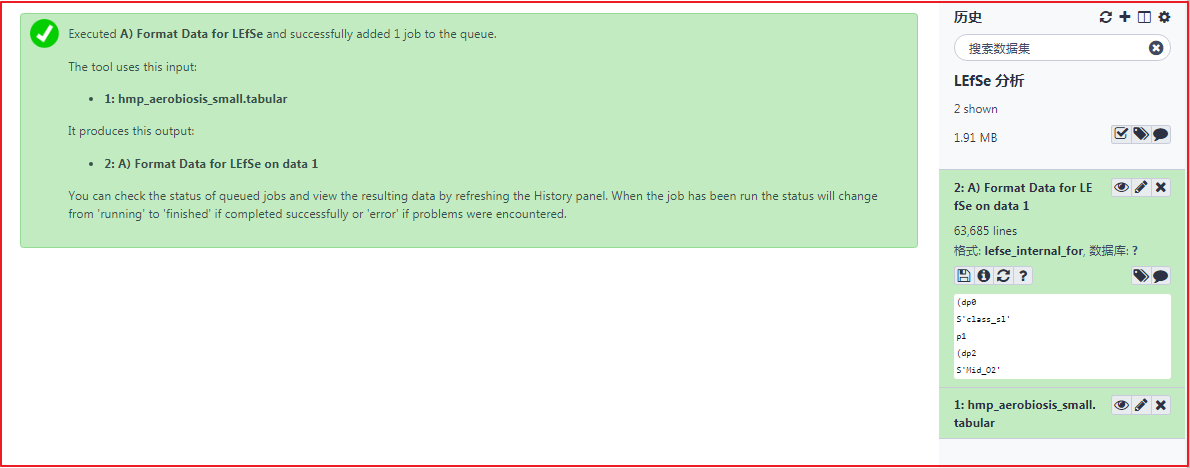
A. Format Data for LEfSe¶
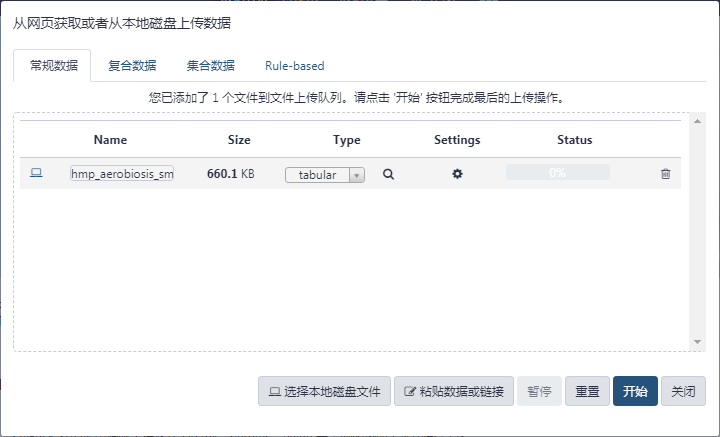
第一步,上传数据。
点击 Galaxy 的左边工具栏 "获取数据" → "数据上传" → "选择本地磁盘文件" → hmp_aerobiosis_small.tabular → 设置 Type: tabular → "开始"。
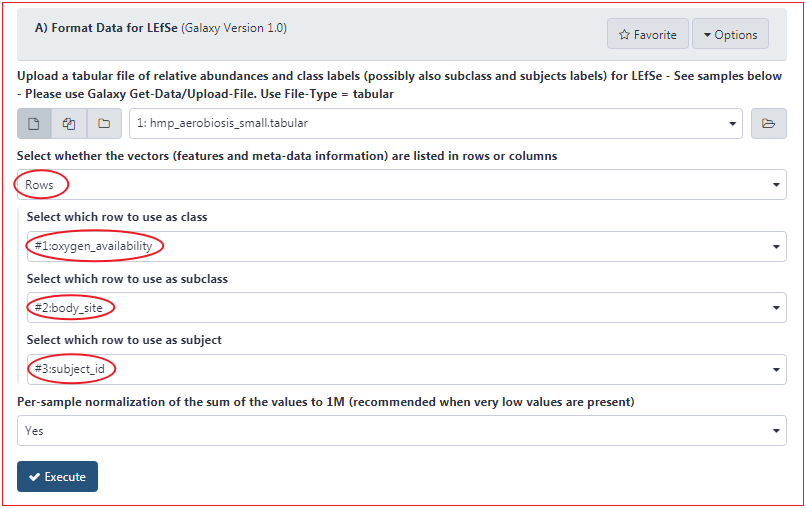
第二步,设置参数,提交任务。
点击 Galaxy 中 LEfSe 分析下的 "A) Format Data for LEfSe",选择第一步输入的数据,设置参数如下。
第三步,查看结果。
任务完成,在右侧历史栏可以看到生成的结果 "2: A) Format Data for LEfSe on data 1"。
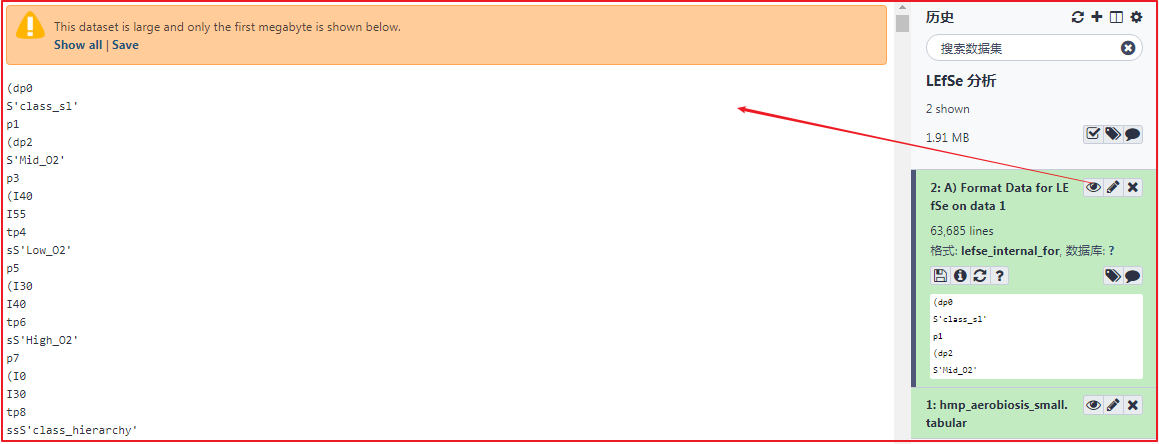
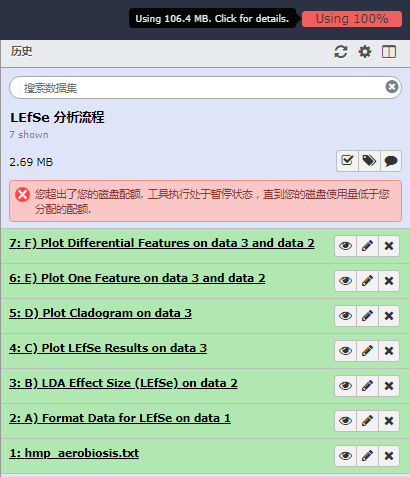
B. LDA Effect Size (LEfSe)¶
第一步,选择数据,设置参数。
点击 Galaxy 中 LEfSe 分析下的 "B) LDA Effect Size (LEfSe)",选择 A 的结果数据,设置参数如下。
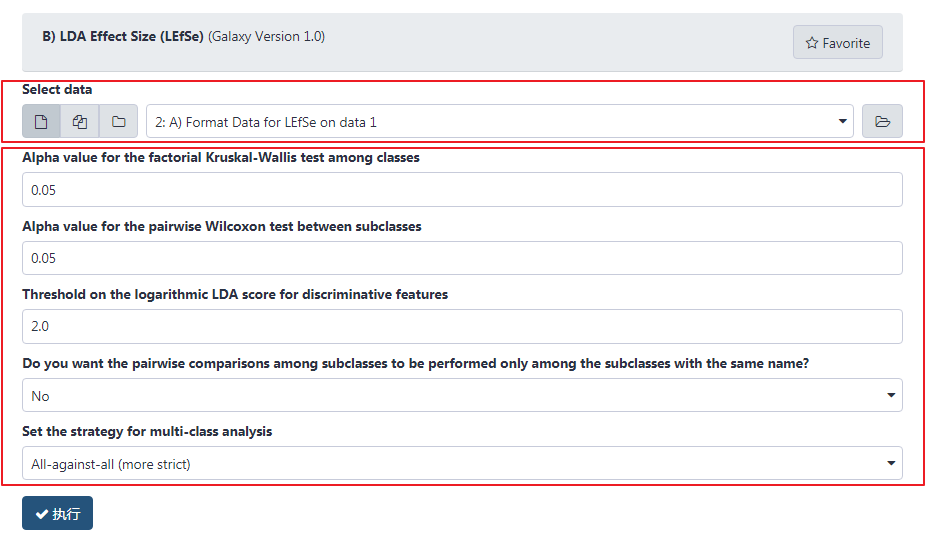
第二步,提交任务,查看结果。
点击 "执行" 提交任务。任务完成,在右侧历史栏可以看到生成的结果 "3: B) LDA Effect Size (LEfSe) on data 2",点击眼睛图标可以到如下结果详细信息。

C. Plot LEfSe Results¶
第一步,选择数据,设置参数。
点击 Galaxy 中 LEfSe 分析下的 "C)Plot LEfSe Results",选择 B 的结果数据,设置参数如下。
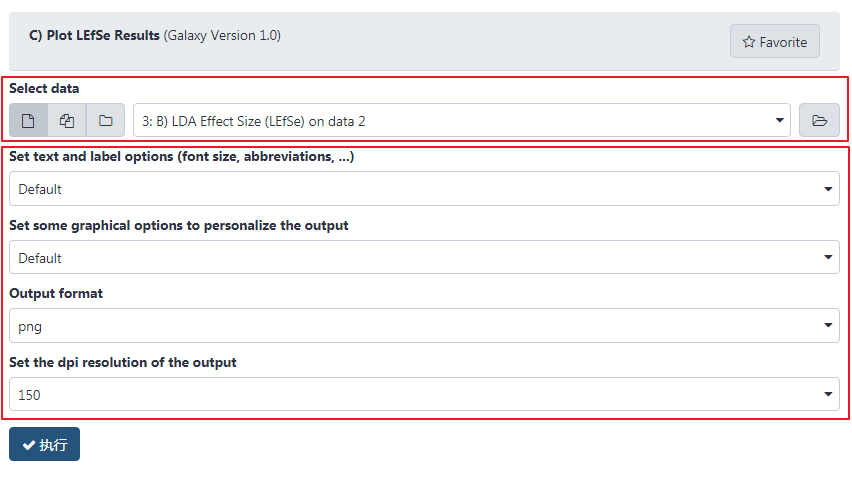
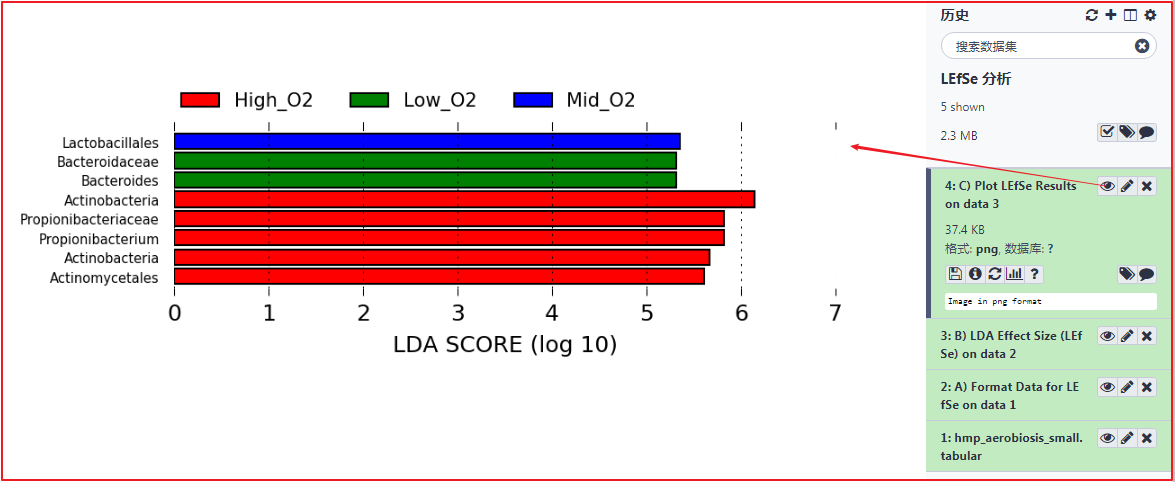
第二步,提交任务,查看结果。
点击 "执行",提交任务。任务完成,在右侧历史栏可以看到生成的结果 "4: C) Plot LEfSe Results on data 3",点击眼睛图标可以到生成的结果。
D. Plot Cladogram¶
第一步,选择数据,设置参数。
点击 Galaxy 中 LEfSe 分析下的 "D) Plot Cladogram",选择 B 的结果数据,设置参数如下。
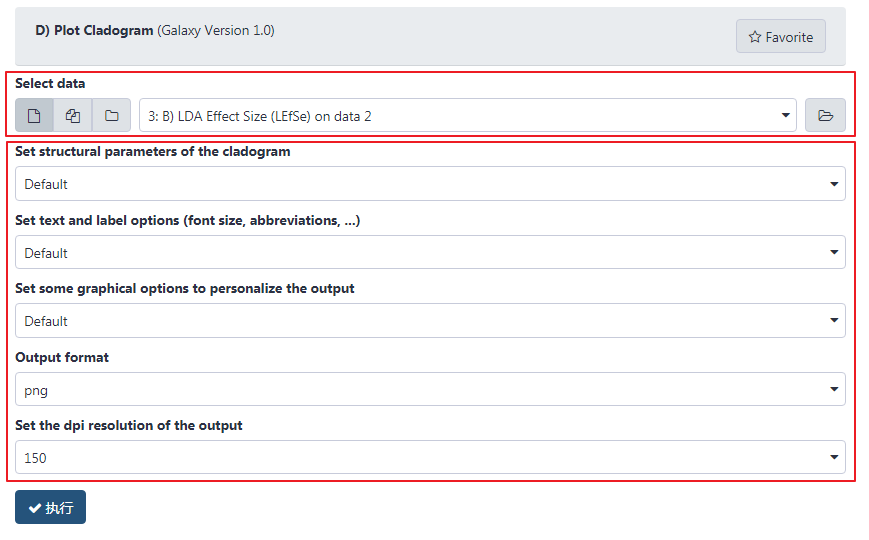 第二步,提交任务,查看结果。
第二步,提交任务,查看结果。
点击 "执行",提交任务。任务完成,在右侧历史栏可以看到生成的结果 "5: D) Plot Cladogram on data 3",点击眼睛图标可以到生成的结果。
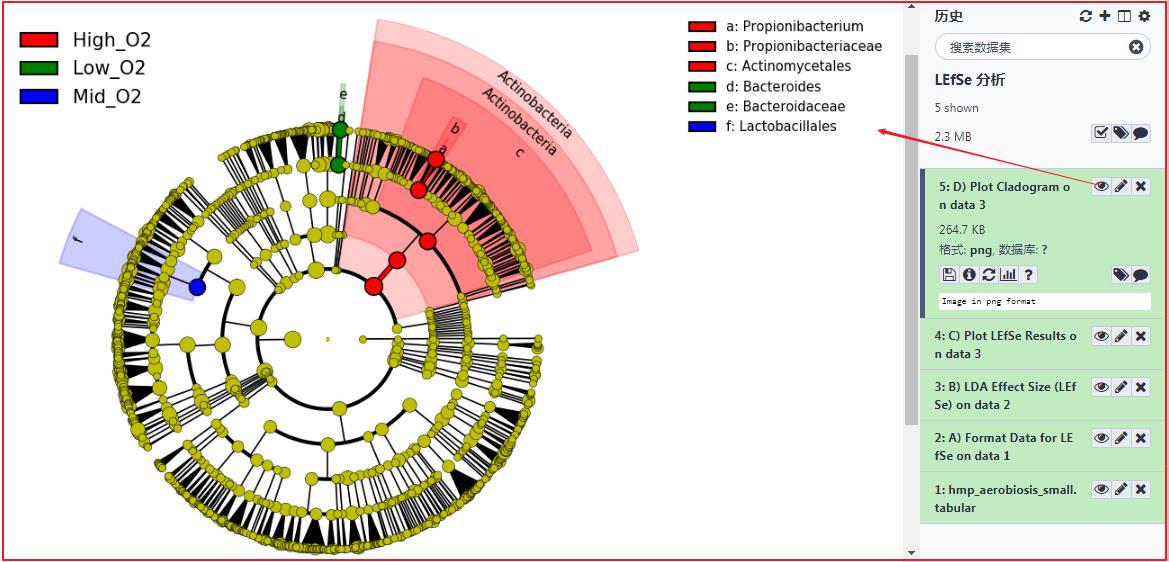
E. Plot One Feature¶
第一步,选择数据,设置参数。
点击 Galaxy 中 LEfSe 分析下的 "E) Plot One Feature",选择 A 和 B 的结果数据,设置参数如下。
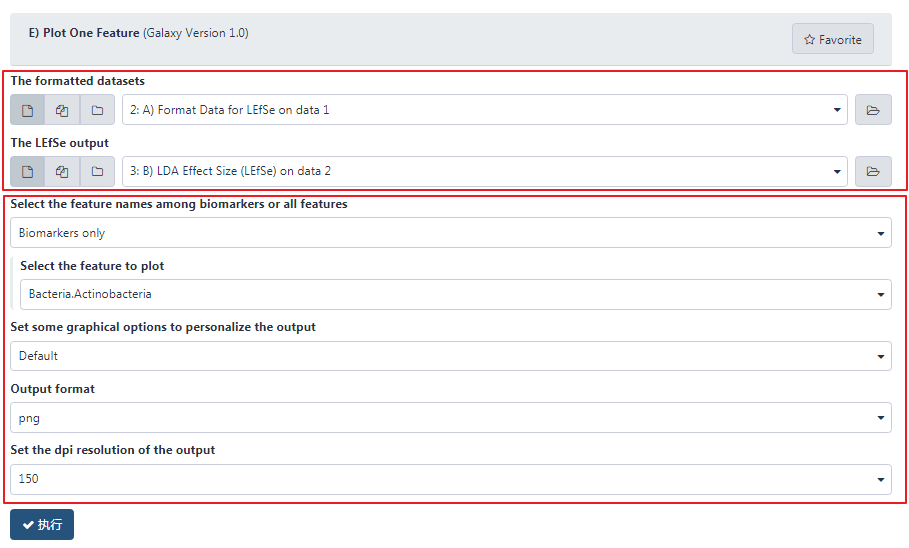
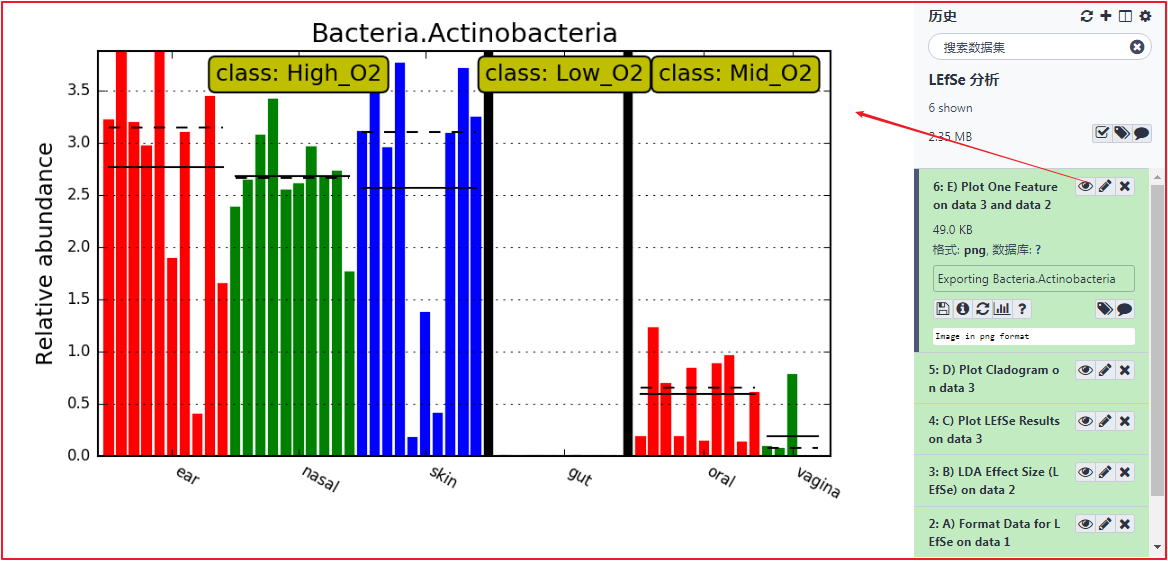
第二步,提交任务,查看结果。
任务完成,在右侧历史栏可以看到生成的结果 "6: E) Plot One Feature on data 3 and data 2",点击眼睛图标可以到生成的结果。
F. Plot Differential Features¶
第一步,选择数据,设置参数。
点击 Galaxy 中 LEfSe 分析下的 "F) Plot Differential Features",选择 A 和 B 的结果数据,设置参数如下。
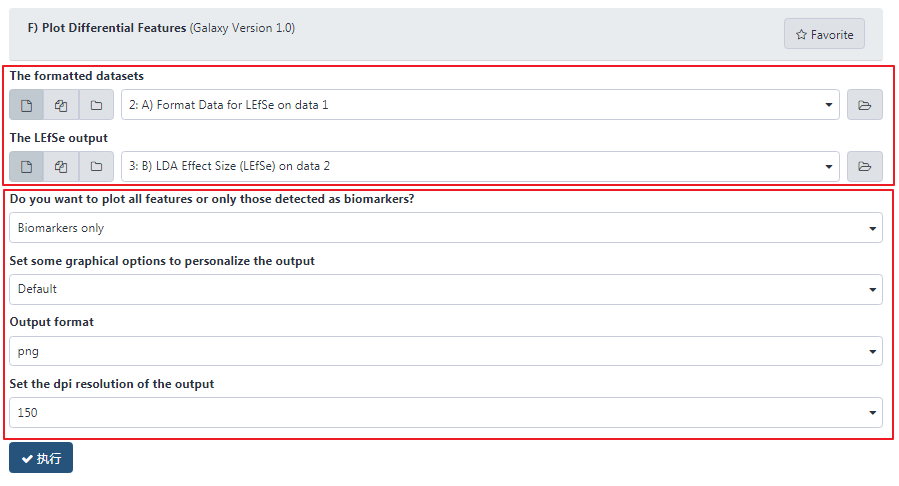
第二步,提交任务,查看结果。
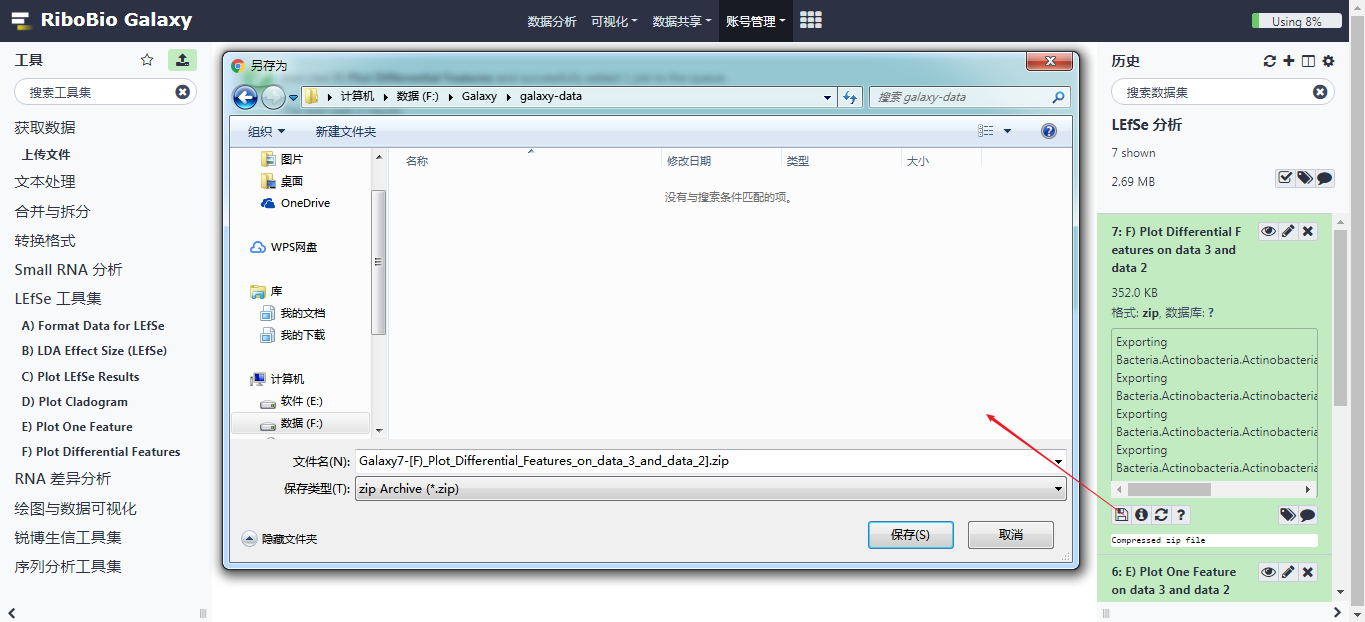
任务完成,在右侧历史栏可以看到生成的结果 "7: F) Plot Differential Features on data 3 and data 2",点击眼睛图片可以下载本次分析的打包文件(*.zip)。
4. 其他工具¶
所有的 RiboBio Galaxy 分析工具,在对应的工具分析参数设置页均提供了非常详细的使用说明以及示例文档,用户可以自行进行查看、测试。这里不再一一赘述。
如在使用过程中有任何的问题,欢迎及时与我们的管理员(galaxy@ribobio.com)进行反馈。
5. 数据管理¶
由于 RiboBio Galaxy 是由锐博内部服务器提供支持,存储和计算资源都很有限,因此我们规定了每个注册用户默认 5.0GB 磁盘容量配额。如果发现您的磁盘已经超额,请注意个人的数据清理。
在开始数据清理前,先了解一下几个相关名词。
5.1 删除历史数据¶
对于整个历史级别的数据删除。
-
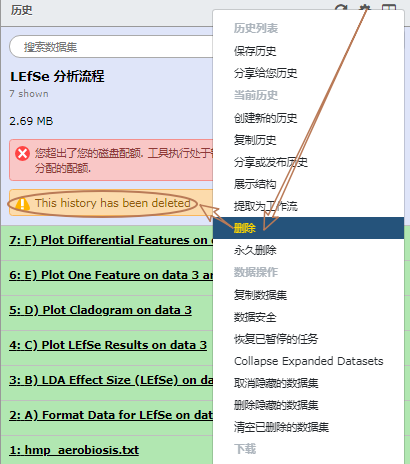
删除:Really delete the current history. 将当前历史的所有数据标记为删除(真正的数据会继续保留在磁盘上),只会在当前历史头部显示数据已删除。
-
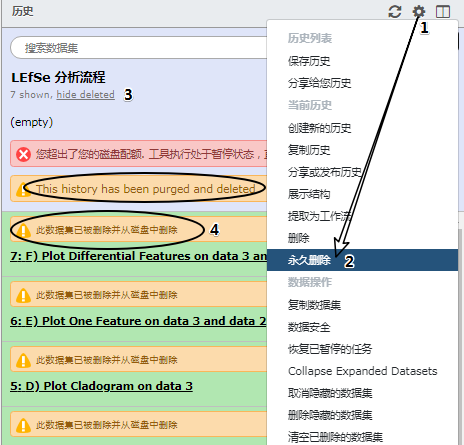
永久删除:Really delete the current history permanently? This cannot be undone. 永久删除数据,会把当前历史下的所有数据从磁盘上清除掉,而且数据无法恢复!
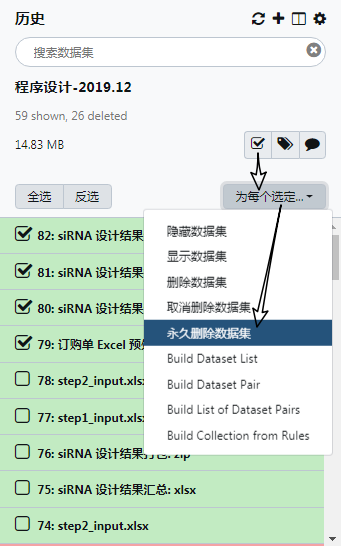
5.2 删除数据集¶
对于数据集级别的数据删除。
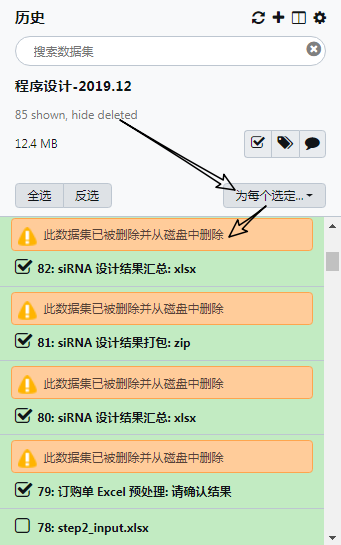
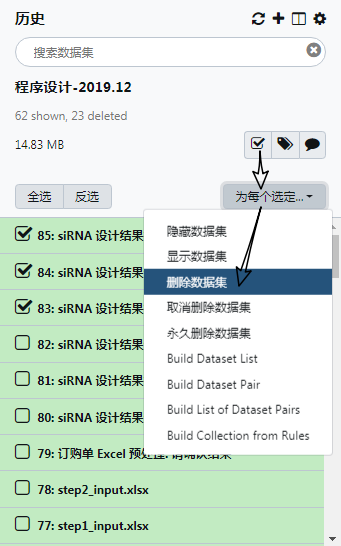
Galaxy 里面数据集的概念,就是指我们在历史栏中看到的一个个的数据,例如下面截图中的 81、82 、... 都是数据集。每个数据集后面都都三个小图标,分别表示查看当前数据集、编辑当前数据集的属性、将当前数据集标记为删除。
-
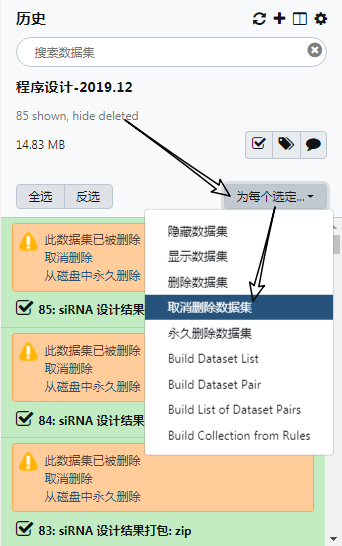
删除数据集:这个跟每个数据集后面的 "X" 删除图标的效果是一样,唯一区别在于这是对多个数据集进行操作。删除数据集只是把数据集标记为删除,真正的数据会继续保留在磁盘上。而且,数据集级别的标记删除是可以恢复的。
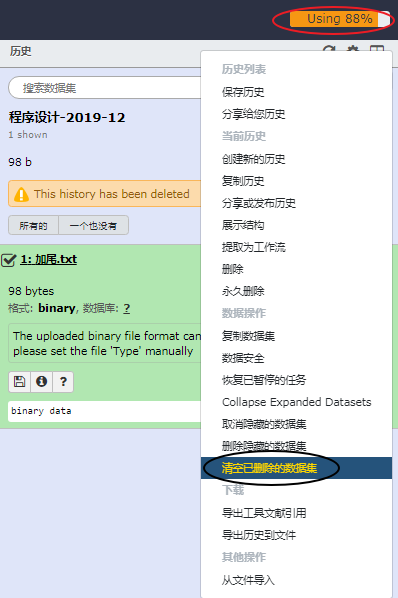
5.3 清空已删除的数据集¶
清空已删除的数据集(Really delete all deleted datasets permanently. This cannot be undone.),本操作会把你 所有历史 中已经标注为 删除 的数据全部 从磁盘中清除,而且数据清除后 不可恢复! 所以,我们平时所说的数据清理,指的就是数据已经从磁盘中被清除掉。而且只有数据从磁盘中被清除掉后,你的磁盘使用量才会被释放!
6. 支持服务¶
我们基于免费的有限资源提供本平台服务。
在使用本平台的过程中,如果您有任何疑问或者建议,欢迎随时通过 galaxy@ribobio.com 与我们反馈。
温馨提示
邮箱 galaxy@ribobio.com 专门用于接收本平台所有用户在使用及注册过程中遇到的相关问题,我们承诺不会通过此邮箱向任何用户主动发送任何信息。